¿Te has puesto alguna vez a pensar en la cantidad de plástico que usas en tu vida diaria? Más específicamente, ¿la cantidad de plástico que desechas cada día? ¿Cuántas botellas de agua o de refresco por semana? ¿Cuántos vasitos, bolsas, contenedores o envolturas desechables? Pues bien, la mayor parte de estos objetos están hechos de Tereftalato de polietileno, mejor conocido como PET, el tipo de plástico más producido del mundo.
De obesidad en perros y bacterias intestinales
 En el mundo existen 500 millones de personas obesas, mismas que presentan la diversidad más pobre de comunidades bacterianas en su aparato digestivo. Dicha relación aplica también para los perros obesos.
Esta conclusión se obtuvo luego de estudiar a perros conocidos como “Beagle”. Un grupo de investigadores de instituciones coreanos y estadounidenses alimentaron a cierto número de perros con cantidades ilimitadas de comida durante seis meses. En este tiempo, cada animal ganó más de siete kilos, que fue cerca del 67% de su peso inicial. Otro número de perros fue alimentado con porciones controladas de alimento, y éstos no ganaron peso.
En el mundo existen 500 millones de personas obesas, mismas que presentan la diversidad más pobre de comunidades bacterianas en su aparato digestivo. Dicha relación aplica también para los perros obesos.
Esta conclusión se obtuvo luego de estudiar a perros conocidos como “Beagle”. Un grupo de investigadores de instituciones coreanos y estadounidenses alimentaron a cierto número de perros con cantidades ilimitadas de comida durante seis meses. En este tiempo, cada animal ganó más de siete kilos, que fue cerca del 67% de su peso inicial. Otro número de perros fue alimentado con porciones controladas de alimento, y éstos no ganaron peso.
Al examinar los restos fecales de todos los perros, los investigadores observaron que el sistema digestivo de aquellos obesos contenían una diversidad pobre comparada con la de los perros en su peso. Además, cada grupo de animales tuvo representado diferentes tipos de bacterias.
Los investigadores mencionan que la abundancia diferencial de microorganismos entre los dos perros puede estar relacionada con el aumento de peso, algo que ya se ha visto con ratones.
Fuentes:
Artículo original | Nota de Science | Nota Original en el Blog de Historias Cienciacionales | Imagen tomada de este sitio.
Los besos saben a miles de bacterias
 Un beso de los buenos ayuda a enamorarse de la misma manera en que uno se queda dormido: lento y todo al mismo tiempo. Con los besos, como dijera Cortázar, basta con cerrar los ojos para deshacerlo todo y recomenzar.
Mientras tocamos el borde de tu boca con un dedo, te contamos que por cada beso largo, transfieres cerca de ochenta millones de bacterias a la boca de tu pareja.
Un beso de los buenos ayuda a enamorarse de la misma manera en que uno se queda dormido: lento y todo al mismo tiempo. Con los besos, como dijera Cortázar, basta con cerrar los ojos para deshacerlo todo y recomenzar.
Mientras tocamos el borde de tu boca con un dedo, te contamos que por cada beso largo, transfieres cerca de ochenta millones de bacterias a la boca de tu pareja.
Llegar a dicha conclusión es complicado, debido a que si los científicos toman muestras de las bocas de los dos en cuestión antes y después del ósculo, la diferencia en la composición bacteriana es pequeña. Hay dos posibles razones para esto. La primera es que, después de ya haberse dado muchos besos, sus bocas se vuelven hogar de las mismas poblaciones bacterianas. La segunda es que, de tener una relación, sus similares estilos de vida influyen en el tipo de habitantes bucales.
Una manera de analizar el número de bacterias compartidas después de un beso es darle de comer a uno de los dos un yogurt con bacterias inusuales en la boca. Esto fue lo que un grupo de investigadores de los Países Bajos practicó con veintiún parejas en un zoológico de Ámsterdam, y cuyo análisis mostró este número de microorganismos transferidos.
Pese a que ochenta millones suena a un número grande de bacterias, el número se queda pequeño cuando lo comparamos con los cerca de mil millones de estos microorganismos que habitan nuestra boca.
Finalmente, hay más que una sola saliva y un solo sabor a fruta madura: para ser precisos, ochenta mil bacterias después de jugar al cíclope.
Fuentes:
Artículo original | Nota de Science | Artículo original en el Blog de Historias Cienciacionales | Imagen tomada de este sitio
Tu ropa de ejercicio es la que te hace apestar.
 ¡Puaj! Esa peste repugnante que emana de tu habitación, te informa que es momento de lavar tu ropa deportiva. Lo peor de todo es que no puedes culpar a nadie más por tan asquerosa situación. En realidad, sí puedes.
Las bacterias que se localizan en nuestra piel y ropa son las responsables de darle el olor a nuestro sudor. Sin embargo, hay otro factor que determina si la magnitud de la fragancia que despide tu cesto de ropa sucia es suficiente para matar a alguien de asfixia: los textiles.
¡Puaj! Esa peste repugnante que emana de tu habitación, te informa que es momento de lavar tu ropa deportiva. Lo peor de todo es que no puedes culpar a nadie más por tan asquerosa situación. En realidad, sí puedes.
Las bacterias que se localizan en nuestra piel y ropa son las responsables de darle el olor a nuestro sudor. Sin embargo, hay otro factor que determina si la magnitud de la fragancia que despide tu cesto de ropa sucia es suficiente para matar a alguien de asfixia: los textiles.
El crecimiento bacteriano es diferente en algodón y en poliéster. Si se realiza ejercicio intenso usando cualquiera de estas dos telas y se les deja reposar por un día –para que las bacterias transformen las moléculas de nuestro sudor en potentes bombas olorosas- se obtendrá una tela sintética de poliéster mucho más apestosa que la de algodón.
Dicha asquerosa experiencia fue llevada a cabo por diferentes investigadores belgas y neerlandeses. Una vez que olieron los dos tipos de telas, observaron bajo el microscopio que el poliéster no absorbe la humedad, sino que la almacena entre las fibras sintéticas.
Como si no hubiera sido suficiente oler la ropa de veintiséis participantes, los investigadores ahora trabajan en determinar la razón por la que el poliéster es un refugio para las bacterias. Y mientras tanto, ellos recomiendan que para no ofender con su olor letal, los deportistas cambien sus ropas sintéticas por unas de algodón.
Fuentes:
Artículo de libre acceso| Nota de Science | Nota original en el Blog de Historias Cienciacionales| Imagen tomada de este sitio
Un vaso de pulque teotihuacano para llenar la panza.
 En una de las cartas dirigidas al rey Carlos I de España, Hernán Cortés escribe que “El pulque es un vino que ellos beben”. Para cuando el conquistador español llegó al actual México, esta bebida alcohólica era consumida por los aztecas. Sin embargo, antropólogos y arqueólogos han tenido poco claro si el pulque era popular en Teotihuacan.
El pulque, una bebida alcohólica preparada a partir de la fermentación de la savia que se extrae del corazón del agave maduro, se asocia fácilmente con celebraciones y rituales, aunque también es una fuente importante de nutrientes y agua.
En una de las cartas dirigidas al rey Carlos I de España, Hernán Cortés escribe que “El pulque es un vino que ellos beben”. Para cuando el conquistador español llegó al actual México, esta bebida alcohólica era consumida por los aztecas. Sin embargo, antropólogos y arqueólogos han tenido poco claro si el pulque era popular en Teotihuacan.
El pulque, una bebida alcohólica preparada a partir de la fermentación de la savia que se extrae del corazón del agave maduro, se asocia fácilmente con celebraciones y rituales, aunque también es una fuente importante de nutrientes y agua.
Un grupo de investigadores de diferentes centros de estudio, como el Instituto Nacional de Antropología e Historia, realizaron excavaciones en Teotihuacan con el objetivo de analizar vasijas de cerámica utilizadas para almacenar el pulque y examinar si los habitantes de la ciudad más grande de la Mesoamérica precolombina consumían pulque.
El estudio de los residuos químicos mostró que catorce vasijas dieron positivo para ingredientes clave en la producción del pulque, como es la bacteria llamada Zymomonas mobilis.
Las vasijas, que datan de entre los años 200 y 550 de esta era, dan evidencia de la producción de alcohol más temprana en Mesoamérica. Además, los autores del trabajo proponen que el pulque que alguna vez estuvo contenido en éstas, sirvió como un suplemento en la dieta de los teotihuacanos, pues un solo vaso les proveía el sentimiento de saciedad en las épocas en que el alimento escaseaba.
El método novedoso de esta investigación permitirá identificar en futuros estudios antropológicos y arqueológicos -con un alto grado de eficiencia- a bacterias fermentadoras en diferentes bebidas alcohólicas, como es la cidra, el vino o la cerveza.
————————————————-
Artículo original | Nota de Science| Artículo Original en el Blog de Historias Cienciacionales | Imagen del archivo Casasola llamada “El brindis”. Tomada de este sitio
Bacterias que producen un combustible fósil alternativo.
 Investigadores del Imperial College en Londres han logrado modificar genéticamente a una bacteria para que produzca propano, un combustible fósil. “Los combustibles fósiles son un recurso finito y conforme nuestra población sigue creciendo tendremos que recurrir a nuevas formas de cumplir con las crecientes demandas de energía,” comenta Patrik Jones, el coordinador del estudio, para el sitio de noticias de su universidad.
Investigadores del Imperial College en Londres han logrado modificar genéticamente a una bacteria para que produzca propano, un combustible fósil. “Los combustibles fósiles son un recurso finito y conforme nuestra población sigue creciendo tendremos que recurrir a nuevas formas de cumplir con las crecientes demandas de energía,” comenta Patrik Jones, el coordinador del estudio, para el sitio de noticias de su universidad.
Jones y su equipo modificaron el metabolismo de la bacteria Escherichia coli (vieja conocida de la ciencia y de los sistemas digestivos humanos): aprovechan la forma en que la bacteria produce ácidos grasos y los integra en su membrana celular, para agregar algunas enzimas al proceso y terminar con un producto diferente, el propano.
Por ahora, el estudio sólo logró producir una cantidad muy pequeña, insuficiente para comenzar a generarla a nivel industrial, pero el propano conseguido es prácticamente igual al que se usa en la industria. “Aunque esta investigación está en una etapa muy temprana, nuestro estudio proporciona un método de producción renovable de un combustible que anteriormente sólo era accesible a partir de reservas fósiles”, agrega Jones.
Además, los investigadores comentan que siguiendo esta línea de investigación podrían llegar a la producción de combustibles a partir de la energía solar: usando bacterias fotosintéticas en lugar de E. coli.
Fuentes:
Nota fuente en ScienceDaily | Artículo original en Nature | Nota original en el Blog de Historias Cienciacionales | Imagen
Gracias, bacterias, por hacer saludable el chocolate
 Cuando el novio de la Cuca le dijo que ya no quería estar con ella, se puso a comer chocolate oscuro como si no hubiera mañana. La mujer estaba incontrolablemente triste, con el corazón roto. Pero pregúntenle qué tan saludable estaba su cuerpo por las cantidades industriales de este alimento que se embuchaba.
Cuando el novio de la Cuca le dijo que ya no quería estar con ella, se puso a comer chocolate oscuro como si no hubiera mañana. La mujer estaba incontrolablemente triste, con el corazón roto. Pero pregúntenle qué tan saludable estaba su cuerpo por las cantidades industriales de este alimento que se embuchaba.
Su sistema circulatorio estaba sano y tenía una presión sanguínea envidiada por cualquier hipertenso. Los niveles de colesterol le bajaron al punto en que su médico de cabecera estaba orgulloso de ella. Y claro, el poder revitalizante del chocolate la ponía más contenta. Vamos, que el chocolate salvó a la Cuca.
Ahora, la Cuca tiene una razón más para seguir comiendo chocolate oscuro. Resulta que las bacterias de nuestro sistema digestivo rompen los compuestos largos del chocolate en moléculas más pequeñas que el cuerpo puede absorber fácilmente y que pueden frenar la inflamación perjudicial. Así que debemos agradecer a nuestras bacterias por ayudarnos a obtener los beneficios de este alimento.
Las bacterias obtienen un festín con el chocolate oscuro que ingerimos. Después, lo fermentan, generando así los compuestos antiinflamatorios. Para el caso del tejido cardiovascular, estas moléculas promueven que las células se mantengan sanas y el riesgo de sufrir un infarto se reduzca. De ahí que el cardiólogo envidiara el corazón de la Cuca, por más roto que estuviera.
Incluso, si la Cuca llegara a combinar el chocolate oscuro con probióticos –sustancias que simulan la actividad de las bacterias digestivas–, el beneficio sería mayor. Esto se debe a que el número de bacterias que realizan este proceso aumenta y reduce los efectos de aquellas especies de bacterias que causan gases, hinchazón, diarrea o estreñimiento.
Actualmente, la Cuca tiene un corazón en proceso de sanación. También carga con un chocolate extra en la bolsa para usarlo en caso de emergencia pues, según ella, los corazones rotos son a veces impredecibles. ________________
Bibliografía:
Este trabajo fue presentado en la Reunión y Exposición Nacional de la Sociedad Estadounidense de Química (ACS) número 247, en Dallas. Aquí les dejamos el perfil de John Finley, investigador principal del trabajo aquí mencionado
Nota original en Science Daily| Artículo original en Science| Nota en el blog de Historias Cienciacionales
Doce universos bacterianos que no han dejado de evolucionar tras 25 años (ni lo harán)
![Richard Lenski y algunas de las generaciones de bacterias congeladas (aunque están empezando a descongelarse y tal vez por eso no se le ve tan feliz en la foto). A la derecha, uno de los matraces donde crecen y se reproducen las bacterias de Lenski, celebrando los 25 años de experimento (probablemente después de una buena dosis de alcohol). [NPR.org]](http://static1.squarespace.com/static/59057732b3db2ba7ea01fbb3/590978153f4058298d33edb4/590978a53f4058298d34109e/1493792933691/universosbacterianos-300x268.jpg?format=original) ¿Qué estabas haciendo el 24 de febrero de 1988? Así fuera babear tu babero, aprender a gatear, aprender a leer, decir tu primera grosería o coexistir simultáneamente como espermatozoide y óvulo en el interior de tus padres, seguro nada de eso ha sido tan importante para la ciencia como lo que ocurrió en el laboratorio de Richard Lenski en la Universidad Estatal de Michigan. Ese año, a Lenski se le ocurrió abordar una pregunta realmente filosófica sobre la evolución biológica de la manera menos teórica posible. La pregunta era "¿puede la evolución producir dos veces el mismo resultado?"; el método elegido fue un experimento.
Todos sabemos que los seres vivos evolucionan a lo largo de las generaciones y que mientras más generaciones pasan más se notan sus efectos. Pensando en esto, Lenski y sus estudiantes eligieron bacterias de la especie Escherichia coli, que en condiciones de laboratorio engendran una nueva generación más o menos cada hora. Así podrían seguir el cambio en estos microorganismos en el tiempo de vida de un investigador o, en el peor de los casos, en lo que dura un doctorado.
¿Qué estabas haciendo el 24 de febrero de 1988? Así fuera babear tu babero, aprender a gatear, aprender a leer, decir tu primera grosería o coexistir simultáneamente como espermatozoide y óvulo en el interior de tus padres, seguro nada de eso ha sido tan importante para la ciencia como lo que ocurrió en el laboratorio de Richard Lenski en la Universidad Estatal de Michigan. Ese año, a Lenski se le ocurrió abordar una pregunta realmente filosófica sobre la evolución biológica de la manera menos teórica posible. La pregunta era "¿puede la evolución producir dos veces el mismo resultado?"; el método elegido fue un experimento.
Todos sabemos que los seres vivos evolucionan a lo largo de las generaciones y que mientras más generaciones pasan más se notan sus efectos. Pensando en esto, Lenski y sus estudiantes eligieron bacterias de la especie Escherichia coli, que en condiciones de laboratorio engendran una nueva generación más o menos cada hora. Así podrían seguir el cambio en estos microorganismos en el tiempo de vida de un investigador o, en el peor de los casos, en lo que dura un doctorado.
El procedimiento del experimento era sencillo. Pusieron a las bacterias en matraces con medio de cultivo con sólo glucosa como nutriente y las dejaron incubando toda la noche. Al día siguiente, después de que las bacterias agotaran su glucosa y pasaran varias horas con la panza vacía, tomaron un poco del cultivo y lo pasaron a un nuevo matraz con medio fresco y suculento y las dejaron incubando toda la noche en el placer de más comida. Y eso es todo. Suena sencillo, pero lo que distingue al experimento de Lenski y sus colegas de otros son dos cosas: una es que han hecho el mismo procedimiento cada día durante veinticinco años y, dos, que no sólo pusieron un frasco con cultivo de bacterias, sino doce al mismo tiempo. Estos doce cultivos provenían todos de la misma cepa de bacteria; es decir, comenzaron con prácticamente la misma información genética. Lenski y sus colegas querían observar si en cada uno de los doce frascos las bacterias tomaban diferentes caminos evolutivos aun cuando todos tuvieran las mismas condiciones de nutrientes e incluso compartieran la misma charola en la incubadora. Digamos, fue como si hubieran puesto doce universos a correr desde el principio.
A la fecha, el experimento lleva más de 50,000 generaciones de bacterias y el método no ha variado. Lenski y su equipo han conservado muestras de los cultivos cada 500 generaciones. Como las bacterias soportan bien la congelación, se puede meter una muestra de cada linaje al congelador y luego revivirla cuando sea necesario. Así que ahora tienen más de 100 momentos congelados de la historia evolutiva de cada uno de los doce linajes. Los resultados que ha producido el Experimento de Evolución a Largo Plazo (o LTEE por sus siglas en inglés), como Lenski lo ha bautizado, son tantos que valdría la pena revisarlos en un texto más profundo (algo a lo que nos comprometemos desde hoy). Sin embargo, podemos resumir diciendo que por lo menos la pregunta original se pudo contestar desde el principio. Esos doce pueblos de bacterias realmente siguieron distintos caminos evolutivos. Cuando analizaron las diferencias genéticas entre los doce linajes, los investigadores encontraron que, aunque todos los linajes crecían cada vez más rápido, cada uno lo hacía con sus propias mutaciones. Con el tiempo, un linaje incluso desarrolló los medios metabólicos para usar como nutriente un compuesto que ningún otro linaje puede comer. Con el tiempo, algunos linajes dieron lugar a dos diferentes formas de bacterias que coexisten en el mismo frasco. Con el tiempo, algunos linajes comenzaron a mutar más frecuentemente que otros. Lenski no espera saber todo lo que aguarda en los doce futuros de sus frascos, aunque en un artículo publicado esta semana en Science, en coautoría con dos de sus estudiantes, aventura una respuesta.
La forma más directa de saber si las colonias han cambiado a lo largo de las generaciones es ponerlas a competir directamente con sus ancestros y observar cuáles crecen más rápido en el mismo medio. Esto es lo que se entendería, de manera amplia, como adecuación o eficacia en la jerga de los biólogos evolutivos. Las colonias más adecuadas (o, por usar la palabra que le gustaba a Darwin, las más aptas) serían aquellas que se reprodujesen más rápido. Si se quiere, esa capacidad de reproducción se puede ver como un indicador de qué tan bien se están adaptando las bacterias al medio. Luego de comparar las adecuaciones de los doce linajes en su estado actual con las generaciones ancestrales, Michael J. Wiser, uno de los estudiantes de Lenski y primer autor de este estudio, encontró que las generaciones nuevas siempre eran más adecuadas que las anteriores. Es decir, las tasas de reproducción aumentan con el tiempo, así sea un aumento pequeño. Eso no significa que esas bacterias que están evolucionando en un laboratorio de la Universidad Estatal de Michigan llegarán algún día a ser las mejores bacterias del mundo. En realidad, sólo llegarán a ser las mejores bacterias en el mundo de los matraces con medio de cultivo de glucosa. Se están adaptando a su medio en particular y cada linaje lo hace a su modo. Lo que quizá sea más sorprendente es que es probable que nunca dejen de estar adaptándose, a pesar de que su ambiente se mantiene constante. Cuando Wiser generó distintos modelos matemáticos para averiguar cuál predecía mejor la forma en que las bacterias se han hecho cada vez más adecuadas, encontró que los datos se ajustaban mucho mejor a un modelo de crecimiento llamado ley de potencias. En este modelo, el cambio es rápido al inicio, pero luego se vuelve cada vez más lento hasta el punto en el que parece que ya no habrá más cambio; sin embargo, ese punto nunca llega. La gráfica de potencia no tiene límite superior. Wiser dice, para el sitio de noticias de su universidad, que es como estar escalando siempre los últimos pasos del pico de una montaña.
La pregunta que surge enseguida es "¿entonces las bacterias tendrán algún día una adecuación infinita?" ¿Es siquiera eso posible? Sarah Crespi, reportera de la revista Science le hizo esta pregunta a Lenski en una entrevista reciente. Fiel a su tradición, él contestó con otro experimento, pero esta vez imaginario.
Aquí lo parafraseamos: Supongamos que alguien seguirá el LTEE después de que yo muera, y que también alcanzará a generar 50,000 generaciones de bacterias. Y supongamos que ese científico algún día le pasa la estafeta a una nueva generación de científicos, que genera la misma cantidad de generaciones bacterianas, y así y así hasta que el experimento alcance las 50,000 generaciones de científicos. En ese momento, se tendrían 2.5 mil millones de generaciones de bacterias, cada una con mayor adecuación que la anterior. ¿Eso significaría que tendríamos algo como bacterias que se reproducen cada 2 segundos? No. Aunque el modelo predice que habrá aumento constante en la adecuación, la cifra que arroja para la generación 2.5 mil millones es la de una colonia con una adecuación 4.7 veces mayor que la generación original. Esto significa, que se reproducirá en unos 23 minutos, una tasa que se ha observado en otras especies bacterianas, eso sí, sólo cuando hay abundancia de recursos. (Lenski no menciona nada sobre si esos científicos, luego de 50,000 generaciones, casi un millón y medio de años, seguirán siendo humanos o qué. Tal vez basta con que tengan manos y ganas de hacer doctorados.)
El experimento de los doce linajes bacterianos de Lenski todavía tiene muchos frutos que ofrecer. Luego de veinticinco años, se ha convertido en el experimento sobre evolución más importante que se haya hecho. Y para celebrarlo, Historias Cienciacionales te propone una actividad interesante. Si tú también tienes alrededor de 25 años, junta a 11 de tus amigos, preferentemente muy cercanos, (lo mejor sería que hayan vivido en la misma charola de la incubadora), colecten las Escherichia coli que habitan en los intestinos de cada uno (con el método de su preferencia), y échenle un ojo a los genes de esos microorganismos. Seguro encontrarán que las panzas de cada uno de ustedes tiene una historia evolutiva muy diferente que contar (más aún si tus 11 amigos y tú no han vivido en la misma incubadora; recordemos que el experimento de Lenski no ha variado en nada las condiciones). Luego, reporten sus resultados en una revista científica. Tal vez, sólo tal vez, así resarciremos un poco a la ciencia por haber estado babeando, gateando o haciendo la singamia el 24 de febrero de 1988, cuando "el hombre que embotelló la evolución", como se le ha llamado a Lenski, sembró su primera generación de bacterias.
Bibliografía:
El artículo más reciente de Lenski, en coautoría con Wiser | Comunicado de la Universidad de Michigan sobre dicho artículo | Entrevista en podcast para Science | Sitio oficial del laboratorio de Lenski, con mucho contenido educativo y hasta el procedimiento para crear tu propio experimento de evolución a largo plazo.
No eres tú, es tu microbioma
Todo indica que los microbios que forman parte de nuestro organismo pueden influir en nuestra evolución. Esto lo acaban de demostrar investigadores de la Universidad de Vanderbilt en Nashville, específicamente para tres especies de avispas. Si bien cada vez sabemos más sobre los microorganismos que viven en simbiosis con nosotros (colectivamente llamados "microbioma"), su papel en los procesos evolutivos es poco claro. La idea más inquietante al respecto es que la presencia de un microbioma nos convierte en algo más que un individuo. Por dar un ejemplo: en los humanos, el número total de células microbianas es mayor que el número de células de la persona. Somos un colectivo de organismos en coexistencia. Es de esperar que nuestros genomas y los de nuestros inquilinos tengan una estrecha relación. Los científicos han nombrado a esa sociedad como "hologenoma". Así, una de las propuestas más recientes es que el hologenoma, más que el genoma de cada uno de los individuos, puede ser uno de los elementos centrales de la evolución.
Robert M. Brucker y Seth Bordenstein, los autores del estudio, publicado esta semana en Science, analizaron a tres especies de avispas cercanamente relacionadas (del género Nasonia) y a sus microbiomas. Cualquiera de las tres especies era capaz de engendrar híbridos con las otras. Sin embargo, los híbridos de las dos especies más emparentadas tenían un mayor porcentaje de supervivencia, mientras que los híbridos de cualquiera de esas especies con la tercera, más distante evolutivamente hablando, no sobrevivían tan bien. Los científicos encontraron que los microbiomas resultantes en los híbridos no viables eran muy distintos a los de sus padres y resultaban caóticos. Esto explicaba en parte la inviabilidad de los híbridos.
Para probar que los microorganismos asociados eran los responsables de la mortandad de los híbridos, criaron a las avispas en un ambiente estéril y les dieron antibióticos. Para su sorpresa, los híbridos sobrevivían mucho mejor sin los microbios heredados de sus padres, incluso aquéllos que provenían de las especies menos relacionadas. Cuando les devolvían sus microbios asociados, los híbridos, volvían a tener problemas para sobrevivir.
Dado se piensa que uno de los requisitos principales para la especiación es que los híbridos sean inviables, y no siempre se tenían los elementos para explicar esa inviabilidad, el estudio de Brucker y Bordenstein abre nuevos horizontes en el campo. "Nuestros resultados mueven la controversia sobre la evolución hologenómica de una idea a un fenómeno observado", dice Bordenstein para el sitio de noticias de su universidad. "La cuestión ya no es si el hologenoma existe, si no qué tan común es."
Nota de la Universidad de Vanderbilt | Para conocer más del tema, el blog de Bordenstein (en inglés):
Evolution 2012: Que la ciencia funcione
Empecé a escribir esto en Ottawa, Canadá, ahora estoy en mi casa después de varias horas de viaje. Hace unos días eran las últimas horas de Evolution 2012, el congreso sobre evolución que por primera vez conjuntó a las sociedades del estudio de la evolución de Estados Unidos (SSE), Canadá (CSEE) y europea (ESEB). Dos mil quinientos participantes registrados, dos mil quinientas cabezas cuya profesión es estudiar la evolución. Dos mil quinientos científicos que se formaron como biólogos, bioquímicos, matemáticos o computólogos. Dos mil quinientas personas cambiando de salas cada quince minutos, buscando en sus celulares o en sus libretas a cuál plática entrar después. Gente que expuso su trabajo de años en diez diapositivas, estudiantes que discutimos los siguientes pasos de nuestro doctorado de espaldas a un poster con gráficos y letras que resumen nuestros resultados. Foros de discusión. Mucho café.
Trato de sentarme a escribir con la disciplina que se debe. Quiero hacer un recuento de experiencias, pasar en limpio los apuntes, dejar en claro la lista de artículos que tengo que leer y los métodos en los que quiero profundizar, organizar las ideas que discutí con otros estudiantes e investigadores, organizar en mis favoritos los links a diferentes proyectos que me interesaron. Recapitular antes de que todo quede en el conocido olvido de la memoria. Y entonces, como buena procastinadora que soy, decidí escribir esta entrada de blog.
Los biólogos, como el resto de los científicos, tenemos congresos. Reuniones convocadas por la sociedad de estudio de tal o cual campo. Ustedes ya lo saben, yo no lo sabía cuando empecé a estudiar biología. O cuándo menos no tuve claro de qué se trataban hasta que fuí a uno por primera vez. El de Congreso Mexicano de Botánica en Zacatecas, años atrás. Lo comento porque para mí los congresos han sido parte crucial de mi formación. Sé que tal es el caso de tantos otros. Los motivos sobran: una va y se sienta a escuchar un bombardeo de ideas, vistazos a métodos y resultados que aún no se han publicado; una expone su trabajo y otros escuchan y preguntan; y así estudiantes e investigadores quedan inmersos en una atmósfera de retroalimentación que en lo personal considero muy productiva.
Pero eso sobra decirlo, de eso se trata. Si en verdad o no valen el gasto y el viaje, si las cosas van a dirigirse a teleconferencias, si el tamaño importa y si son un reflejo de la geografía de dónde se hace la ciencia son temas más serios que discutiré en otra ocasión. En realidad en esta entrada quiero recapitular un par de sucesos que me gustaron por el puro gusto de contarle al mundo la profesión tan bonita que tenemos y el gozo que es asistir a un congreso.
Empecemos por la diversidad de temas. En una sola mañana y de forma simultánea había pláticas sobre adaptación y genética evolutiva, genética ecológica, comportamiento, filogenética, sistemas de apareamiento y selección sexual, interacción interespecífica y coevolución, por citar unas cuentas. En general una escoge la sala con los temas cercanos a el campo de estudio propio, pero es difícil resistir la curiosidad de entrar, de vez en cuando, a una plática fuera de los estrictos límites de la especialización. Así, por ejemplo, un día había tenido suficiente de genómica y aplicaciones de secuenciación de siguiente generación y decidí entrar a un simposio sobre simbiosis, en particular sobre parásitos que manipulan a su hospedero. Descubrí con asombro la existencia de los gordian worms (o horsehair worms), un nematomórfo (i.e. un tipo de gusano) maravilloso que parasita insectos: por ejemplo crece dentro de un grillo y cuando ha alcanzado la madurez… créanlo o no lo hace cometer un acto suicida: brincar a un cuerpo de agua (lago, río o hasta alberca), donde entonces el gusano parásito puede emerger y continuar con su ciclo de vida en el agua. Aquí un video:
[videoembed type="youtube" align="aligncenter" width="420" height="315" url="http://www.youtube.com/watch?v=Df_iGe_JSzI" id="0"]
De la voz de David Hughes, el orador en cuestión, aprendí que este tipo de interacciones son viejas, existen fósiles que nos hacen pensar que los nematomórfos llevan por lo menos 44 millones de años con tan espectacular forma de parasitismo. De ahí la exposición derivó a un breve recuento de la historia del estudio de estos bichos, con la conclusión de cómo pasó de un sistema anecdótico a métodos ecológicos y genéticos con los que ahora se trata de profundizar en la evolución de estos organismos. La plática era, en realidad, una introducción al resto de las sesiones del simposio, a las cuáles no asistí porque en una sala paralela se comenzó a hablar de cómo poner a prueba modelos demográficos y filogeográficos, esas cosas que yo hago, pues. En fin, entiéndase que había de todo y que más allá de oír en qué va la última investigación del tema propio, los congresos me recuerdan también que la biología sigue creciendo como micelio en la bastedad de direcciones que el mundo natural y sus fenómenos nos pone enfrente. En lo personal, el sólo hecho de saber que bichos como los nematomorfos existen y que alguien se dedica a estudiarlos alimenta mi ánimo.
Creo sin embargo, que lo que da gusto no es sólo que haya gente estudiando este mundo nuestro (bueno, y otros) sino que se está haciendo con buena ciencia. Con el muchas veces lento proceso de poner a prueba hipótesis y llegar a conclusiones basadas en evidencia confiable. Nos guste o no el resultado. Rosie Redfield lo dejó en claro en su muy divertida conferencia magistral. En resumen, esta mujer detectó fallas en los métodos de un artículo publicado en Science, por un grupo de la NASA. El artículo decía haber encontrado bacterias que podían utilizar arsénico como biomolécula e incluso incorporarlo en su ADN en vez de fósforo. De ser cierto, esto hubiera tenido fuertes implicaciones en nuestro entendimiento de los seres vivos. Según Rosie, no fue sólo un problema metodológico de la autora principal, sino una cadena de fallos en todo el resto de las personas involucradas.
Foto de su diapositiva:
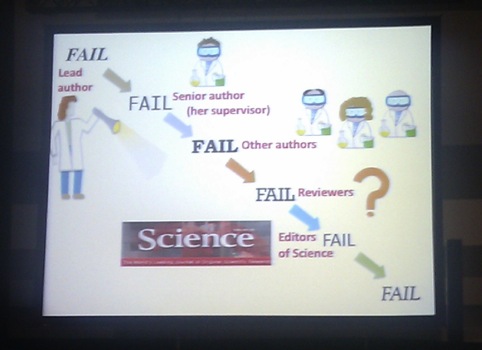
Traducción (de la esquina superior izquierda en dirección de las flechas): Fallo de la autora principal - Fallo del último author (supervisor de la autora principal) - Fallo de otros autores - Fallo de los revisores - Fallo de los editores de Science - Fracaso
Para no hacerles el cuento largo, el artículo ocasionó mucha controversia en la blogósfera de científicos (por cierto que bloguero ya está aceptado por la Real Academía de la Lengua Española), en parte gracias a una entrada y a un tuit de Rosie (el hashtag #arseniclife está bueno). La situación derivó en dos artículos más en Science, uno de ella (y colaboradores) y otro de otro grupo. Básicamente trataron de repetir los experimentos paso a paso, no lo consiguieron y apuntaron la serie de errores y malinterpretaciones del primer estudio. Veremos qué responden los otros. La conclusión al momento: hay bacterias que pueden vivir en arsénico, mas no utilizarlo como fuente alternativa de fósforo ni incorporarlo en su ADN. En otras palabras, después de tantos fallos, la ciencia como proceso sí funcionó:

Traducción: Pero la ciencia (el proceso) funcionó bien.
Me quedé pensando en esa última diapositiva. Se trata de que la ciencia funcione. Pero ya no hablo sólo del método, del basar las conclusiones en evidencia, sino también del resto de los aspectos que hay detrás. De la ciencia como actividad humana. Creo que eso es lo que me llevo del congreso: recordar que a la ciencia la hacen personas, estudiantes e investigadores de todo el mundo, inmersos en nuestros propios problemas, nuestras economías, políticas y pesares. La ciencia como proceso y como actividad humana. ¿De qué depende que funcione (o no)?
[hozbreak]
Acerca del autor
Alicia Mastretta Yanes es Bióloga egresada de la UNAM y actualmente cursa su doctorado en la University of East Anglia, Inglaterra. Su proyecto de doctorado explora la relación entre las características físicas del paisaje y la distribución de la diversidad genética en plantas de alta montaña de México.
Bacteriófagos: Reyes en la virósfera
Cuando escuchamos la palabra virus, la mayoría de nosotros piensa en enfermedades, en parte porque los virus, por ejemplo el VIH, son responsables de enfermedades terribles; de hecho sólo el virus causante de la viruela es responsable de más muertes humanas que cualquier otra enfermedad, se le atribuyen 500 millones de muertes sólo en Europa entre 1400-1800. Otras enfermedades causadas por virus incluyen el resfriado común, la influenza, el SARS y el ébola; sin embargo, la enorme mayoría de los virus no interactúan directamente con los seres humanos, sino que infectan bacterias y tienen una influencia determinante, muchas veces ignorada, en la ecología global del planeta y en la evolución de los seres vivos.
Los virus son organismos que se caracterizan por tener un genoma pequeño, de DNA o RNA, que está protegido por una especie de cápsula hecha de proteínas que se llama cápside. Cuando los virus están fuera de una célula, son partículas inertes (denominadas viriones) que se mueven pasivamente hasta que entran en contacto con una célula susceptible; cuando esto ocurre, las proteínas de la cápside son capaces de reconocer señales en la membrana de la desafortunada célula y provocar la internalización del genoma del virus. Esto puede ocurrir de varias maneras, pero una vez dentro, el programa genético del virus se activa y secuestra la maquinaria celular, redirigiendo el flujo de energía y materiales hacia la producción de nuevos viriones, que eventualmente provocan el colapso de la célula (lisis), dispersando a los viriones y reiniciando el ciclo.
Los bacteriófagos (o simplemente fagos) son virus que infectan células bacterianas, y fueron descubiertos independientemente por Twört en 1915 y D’Herelle en 1917; siendo este último quién acuñó el término bacteriófago: comedor de bacterias. Aunque no podemos verlos, los bacteriófagos son el organismo dominante del planeta. A principios de los noventa se descubrió que en el mar hay 10 veces más viriones que células, y esta proporción ha sido observada en prácticamente todos los ambientes estudiados. Las únicas dos excepciones son el tracto digestivo de pacientes en fase aguda de cólera, y en los pulmones de un paciente con fibrosis quísitica.

En la actualidad se estima que existen 10^31 viriones en el planeta; para darse una idea de lo que eso significa, la población humana es de 7 mil millones (10^9), y el número de estrellas en el universo observable es de sólo 10^24. Además de su gran número, los fagos son increíblemente diversos a nivel genético; aunque pueden clasificarse en sólo 20 grupos de acuerdo a la forma de su cápside, los científicos continúan encontrando nuevos genes cada vez que secuencían el genoma de nuevos fagos; y estudios metagenómicos arrojan que hasta el 80% de la información genética en los bacteriófagos no está relacionada a ningún organismo conocido. Las funciones de la mayoría de estos genes son desconocidas, y se acumulan más rápido de lo que los científicos pueden estudiarlos.
Cuando se encuentran dentro de una célula bacteriana, dos fagos son capaces de recombinar o intercambiar información genética entre ellos, lo que les permite incrementar su diversidad. Sin embargo, cuando algo “falla” en el plan del virus, y su genoma queda atrapado dentro de una bacteria sin ser capaz de reproducirse, la propia bacteria puede tomar genes del fago que le sean útiles, por ejemplo: que le otorguen resistencia a otros fagos, que le permitan eliminar a otras bacterias o -lo más interesante- que le otorguen una nueva función. Desde hace años los científicos habían notado que la capacidad de metabolizar algunos azúcares, o incluso la toxina del cólera fueron adquiridas por las bacterias a través de fagos; pero el ejemplo más importante, tiene que ver con la fotosíntesis. Todos sabemos que las plantas realizan fotosíntesis, pero al menos el 20% de este proceso es realizado por un grupo de bacterias marinas llamadas cianobacterias, mediante un grupo de genes denominados pba. El descubrimiento de que estos genes se encuentran también en los fagos marinos, fue seguido por la observación de que el repertorio de genes pba de una cianobacteria depende de la profundidad en la que vive y de los fagos que la infectan, lo que indica que los bacteriófagos son esenciales para mantener la capacidad fotosintética de los océanos.

El lado depredador de los fagos, aunque temible, también es importante. Retomando al tema de la fotosíntesis, aunque el oxígeno es esencial para la vida, es tóxico en altas concentraciones. Si las cianobacterias pudieran reproducirse libremente, la concentración de oxígeno en la atmósfera se incrementaría terminando con nuestra existencia y la de la vida como la conocemos. Afortunadamente, los fagos matan entre el 20-40% de las bacterias marinas diariamente, manteniendo el equilibrio de los ciclos del oxígeno y del carbono en el planeta. Otra utilidad de los fagos como depredadores, es en el tratamiento de infecciones bacterianas; se trata de una idea muy vieja que fue abandonada cuando los antibióticos se popularizaron a mediados del siglo XX, pero ante la aparición de cada vez más cepas bacterianas resistentes a antibióticos, ha habido un resurgimiento del interés en esta aplicación, principalmente basado en la idea de que los fagos también evolucionan, y por lo tanto pueden responder a las adaptaciones de las bacterias.
Un último punto que me gustaría abordar es el de los bacteriófagos como modelos científicos. Gracias a su simplicidad, abundancia, diversidad y fácil manipulación, los fagos son modelos excepcionales y han estado en el frontispicio de la biología desde su descubrimiento. Gracias al estudio de los fagos: se zanjó en definitiva la polémica sobre la naturaleza del material genético; se descubrió el papel del mRNA en el flujo de la información genética; se lograron elucidar los mecanismos básicos de regulación de la expresión genética y de la replicación del DNA; en los fagos se mapeó el primer gen, se descubrieron las enzimas de restricción y las modificaciones epigenéticas; y los primero genomas secuenciados también fueron de bacteriófagos.
Los virus son mucho más que sólo agentes causantes de enfermedades. Aquí he expuesto sólo la punta del iceberg de la virósfera (el mundo de los virus). Espero haber despertado su interés y agradezco a Más Ciencia por México por invitarme a participar, y no olviden visitar mi blog (link abajo).
Acerca del Autor: Sur Herrera Paredes es egresado de la Licenciatura en Ciencias Genómicas de la UNAM. Actualmente estudia el doctorado en Bioninformática y Biología Computacional en la Universidad de Carolina del Norte, EUA. Además mantiene el blog de divulgación La Ciencia explicada.
Referencias: Rohwer & Vega Thurber. “Viruses manipulate the marine environment” (2009). Nature 459, 207-212. Sharon et al. “Viral photosynthetic reaction center genes and transcripts in the marine environment” (2007). The ISME Journal 1, 492-501. Zimmer. “A planet of viruses” (2011). University Of Chicago Press, 1st edition.





