¿Sabías que las células de nuestro cuerpo saben cuándo es de día y cuándo es de noche? Este fenómeno se llama “ritmo circadiano” y es de gran importancia para regular desde procesos a nivel celular hasta el comportamiento de los seres vivos.
El origen aviar de la gripe española
 Hace casi cien años ocurrió la pandemia más devastadora de la historia de la humanidad. Infectó a 500 millones de personas alrededor del mundo y acabó, al menos, con 50 millones de vidas (aunque hay quienes piensan que esta cifra se eleva hasta los 100 millones). El acontecimiento recibió el nombre de gripe española, y su causante fue un virus.
Hace casi cien años ocurrió la pandemia más devastadora de la historia de la humanidad. Infectó a 500 millones de personas alrededor del mundo y acabó, al menos, con 50 millones de vidas (aunque hay quienes piensan que esta cifra se eleva hasta los 100 millones). El acontecimiento recibió el nombre de gripe española, y su causante fue un virus.
Desde entonces, el responsable de la pandemia de la influenza ha estado bajo el ojo de muchas investigaciones que buscan responder tantas interrogantes planteadas como sea posible. Ahora, el origen de la gripe española es más claro, en tanto que se han estudiado sus relaciones evolutivas con otros virus.
En 2005, un estudio del material genético de tejidos de pacientes que fallecieron durante la gripe española sugirió que había una alta probabilidad de que el origen del virus causante de tantas muertes humanas fuera aviar. Sin embargo, cuatro años después, otro trabajo mostró que los genes virales circularon entre humanos y cerdos por al menos dos o quince años antes de que la pandemia ocurriera, y se combinaron para generar un virus letal.
Recientemente, tres investigadores de diferentes instituciones publicaron un trabajo en el que analizaron más de 80 mil secuencias genéticas de virus de la gripe provenientes de humanos, aves, caballos, cerdos y murciélagos. Gracias a esto, ha sido posible conocer los orígenes del virus A de la influenza y se ha podido observar cómo ha evolucionado a lo largo de dos siglos, pasando a través de diferentes animales. Así, se ha concluido que el virus de la gripe española provino de aves domésticas y silvestres de Norteamérica, y no a partir de la mezcla de virus humanos y porcinos, como se pensaba.
Para poder llegar a dicho resultado, los científicos partieron de un supuesto: los cambios genéticos se acumulan en el tiempo a una tasa confiable, como el segundero de un reloj. De ahí que a este modelo, que permite unir piezas del rompecabezas de las relaciones evolutivas entre organismos y sus mutaciones en el tiempo, se le conozca como “reloj molecular”.
Algunos modelos asumen que las tasas evolutivas moleculares son aproximadamente iguales pero, en el caso de la influenza, existe la evidencia de que evoluciona a diferentes velocidades en cada uno de sus hospederos. Esto se sabe porque se ha visto que la tasa de cambio es más rápida en aves que en caballos, por ejemplo.
El análisis de este reciente trabajo también reveló que existe un ancestro común para casi todas las gripes de origen aviar con el virus H7N7, mismo que también fulminó a caballos y mulas en Norteamérica, pero en 1872. Esta panzootia comenzó en Canadá y se dispersó rápidamente hacia el sur y oeste. La transmisión entre humanos y caballos parece ser clave para algunas epidemias, algo que mueve la mirada hacia un origen equino. Si esto fuera cierto, cambiaría el dogma de aves a caballos.
Bibliografía
Nota Fuente en Nature | Artículo sobre influenza aviar moderna en Nature (2014) | Artículo sobre influenza aviar de 1918 en Nature | Artículo sobre influenza aviar en PNAS| Artículo original en el Blog de Historias Cienciacionales | Imagen
Tragedias prehistóricas
 Cada pasillo, rincón o pared del Museo Geológico de la Provincia de Anhui, en China, susurra miles de historias. Historias antiguas, todas ellas extraordinarias: unas tristes, otras violentas y, algunas más, conmovedoras. Con más de dos mil piezas fósiles recolectadas, el museo recopila las voces extintas de unos pocos animales que pisaron el suelo o nadaron en las aguas de una Tierra pasada.
Cada pasillo, rincón o pared del Museo Geológico de la Provincia de Anhui, en China, susurra miles de historias. Historias antiguas, todas ellas extraordinarias: unas tristes, otras violentas y, algunas más, conmovedoras. Con más de dos mil piezas fósiles recolectadas, el museo recopila las voces extintas de unos pocos animales que pisaron el suelo o nadaron en las aguas de una Tierra pasada.
De entre el coro de historias, una resuena particularmente fuerte. Sepultada bajo el breve nombre de AGM I-1, a simple vista no parece ser más que un montón de huesos fosilizados, pero si uno observa con atención podrá leer en la roca el trágico final de una familia atrapada en el tiempo.
Hace 248 millones de años, en algún lugar del vasto mar del Mesozoico, una madre murió durante el parto. Tres eran sus hijos. Uno de ellos, el mayor, salió de su cuerpo, nadó unos cuantos centímetros y ya no volvió a aletear. Otro quedó atrapado en su pelvis, en una suerte de limbo natal, con medio cuerpo fuera y otro medio dentro de mamá. El último, el más pequeño, no cruzó jamás la frontera hacia el mundo exterior y se quedó ahí, paciente, en el interior de su progenitora.
Los cuatro aguardaron juntos y en silencio hasta el año 2010 de nuestra era cuando, con la llegada de septiembre, llegó también Ryosuke Motani, un primate homínido obsesionado con los reptiles marinos del Mesozoico y, en especial, con los ictiosaurios. Por esas fechas, Ryosuke y su equipo desenterraron 80 tumbas, cada una de ellas ocupada por algunos de los ictiosauros más antiguos jamás descubiertos. Pero hubo una en especial que captó el interés de los paleontólogos: la AGM I-1. La madre y sus retoños.
Ryosuke no sólo quedó fascinado por la historia que le contaban los restos de aquella familia, sino también porque significan un cambio de paradigma en la evolución: normalmente, la viviparidad (o el desarrollo de los embriones dentro de su madre) es un proceso observado en la gran mayoría de los mamíferos – con algunos ejemplos raros en otros grupos como artrópodos o peces – mientras que en reptiles predomina la costumbre de poner huevos.Hasta hace poco, se creía que la viviparidad había evolucionado en animales acuáticos que después conquistaron la tierra firme. Pero AGM I-1 demuestra algo muy distinto.
En el fósil, todas las crías están orientadas con el rostro hacia afuera – cosa común, pero en los partos terrestres. Comparados con especies marinas actuales (como tiburones, ballenas y delfines), quienes asoman primero la cola al nacer para evitar ahogarse, los ictiosaurios parecen contradecir los supuestos de muchos años. Para Ryosuke y sus colegas, son un claro indicio de que estos reptiles marinos heredaron de sus ancestros terrestres la capacidad de desarrollarse dentro del vientre materno. Esto quiere decir que los primeros animales vivíparos no nadaban en el mar, sino que se arrastraban en los suelos. "A menos", dice Ryosuke, "que nueva evidencia muestre lo contrario".
En los pasillos del Museo Geológico de la Provincia de Anhui, en China, se escucha el murmullo de los fósiles ahí exhibidos. Uno de ellos cuenta la historia de una madre y sus hijos, pero también la de todo un grupo de hembras que salieron del agua sólo para regresar a ella y, en su retorno, se llevaron a sus crías escondidas en el vientre.
-----------------------------------------------------------------------------------------------------------------------------------------
[La imagen, recuperada del artículo de Ryosuke Motani, muestra un código de colores para observar el espécimen AGM I-1. Las vértebras de la madre están en negro; sus costillas, en verde; su aleta y su pelvis, en azul. El recién nacido se encuentra en rojo; el hijo que estaba en proceso de nacer, en amarillo. El cráneo del embrión atrapado dentro es el anaranjado]
Bibliografía:
Artículo Original en PLOS ONE | Artículo en el Blog de Historias Cienciacionales
Del tac tac al tic toc pasando por las lunetas
Tac, tac, tac suena el teclado de mi computadora mientras escribo en la oscuridad. Tac, tac y rio al pensar lo común que es usar “letras y palabras” como metáfora al hablar de ADN. Lo digo como preámbulo a que en este texto también trataré de simplificar conceptos. Pretendo tan sólo (intentar) explicar algunos de los principios más básicos detrás de una pregunta que me hicieron respecto a otra entrada de blog: ¿Cómo puede ocuparse el ADN para estudiar los cambios en la distribución de la biodiversidad? La respuesta llevó a una muy entretenida plática (cerveza en mano y desilusión futbolística en pantalla) que la limitante de espacio no me deja recrear aquí. Conversación que también empezó con la metáfora de las letras y que dio paso a hablar de las herramientas con las que la filogeografía se arma para explorar los procesos históricos por los que han atravesado las especies.
El ADN es una molécula conformada por unidades llamadas nucleótidos que están enlazadas en una especie de cadena. Estas unidades difieren entre sí en una de sus partes, la base nitrogenada, de la cual existen cuatro* tipos: la adenina (A), la guanina (G), la timina (T) y la citosina (C). Los nucleótidos están entrelazados unos con otros en una secuencia que puede leerse de forma lineal (i.e. algo como GATACAATCCATAGC…). El orden en el que se encuentran genera información biológica de forma similar que las letras de este escrito forman palabras y oraciones que dotamos de significado y que pueden formar párrafos sobre un tema en particular. Así, la secuencia de nucleótidos del ADN forma distintos genes, que son las instrucciones de cómo, cuándo y dónde construir diferentes proteínas. Las proteínas son moléculas que forman parte esencial de nuestra estructura y funcionamiento. Ejemplos clásicos: la queratina que conforma las uñas y el cabello; y la insulina que es indispensable para que las células tomen la glucosa de la sangre.
Estoy segura de que los genómicos (personajes enfocados en ciencias genómicas, pues) que habitan este blog sabrán explicar mejor que yo la maravilla de cómo las proteínas y otras biomoléculas forman un ser vivo. Dejémosles la tarea. Mi intención era sólo introducir al ADN y decir que su secuencia forma los genes que a su vez se traducen en proteínas, o en términos formales, que el ADN “codifica” proteínas. Necesitaba tal introducción para poder decir ahora que no todo el ADN codifica para algo, una buena parte, llamada ADN no codificante (creativo nombre) está presente sin que su secuencia produzca proteína alguna. En algunos casos está involucrado en tareas regulatorias, pero en otros puede no tener función (más tarea para los genómicos: explicarnos su origen).
Ahora otro concepto: la mutación es un cambio en la secuencia de ADN y puede ocurrir por errores de copiado durante la replicación del material genético. Las sustituciones (e.g. AGTGCATGC à AGTGCATCG) son un tipo de mutación, pero hay otros. El fenómeno sucede tanto en el ADN codificante como en el no codificante. La diferencia es que la selección natural “puede ver” las mutaciones que ocurren en los genes si éstas implican cambios en la proteína final y “no puede ver” aquellas que no afectan a las proteínas.
Explico aquello de ver. Sucede que las mutaciones pueden resultar ventajosas, por ejemplo hacer que cierta proteína confiera resistencia a un insecticida en el caso de un chapulín, o al contrario, pueden ser perjudiciales y que el individuo incluso muera antes de nacer. La mutación ventajosa tendrá más posibilidades de pasar a la siguiente generación pues gracias a ella el organismo que la carga podría tener más descendencia (los chapulines sobrevivientes a la fumigación podrán reproducirse), mientras que una mutación desventajosa tendrá menos posibilidades. Y esto es, grandísima simplificación de por medio, a lo que los biólogos nos referimos con que la selección natural “ve”. Ahora viene la ceguera. Como ya decía, la secuencia del ADN no codificante no está involucrada con el producto final, la proteína. Una mutación en un fragmento de ADN no codificante no jugará rol alguno en el éxito de la reproducción del organismo que la carga. Para la selección natural es como si no estuviera ahí. Por eso se dice que muchas regiones de ADN no codificante son neutrales o prácticamente neutrales.
Sin embargo, este ADN físicamente sí existe y se transmite de padres a hijos. La razón es simple: durante la replicación del ADN se copian todas las letras porque el proceso involucra ir siguiendo la cadena como si fueran los rieles de la vía de un tren. El que una mutación de este tipo pase o no a la siguiente generación depende de la frecuencia en que se encuentre en la población, en el mismo sentido en que la probabilidad de sacar una luneta verde o una roja depende de cuántas lunetas verdes y rojas contenga la bolsa. A esto se le llama deriva génica. Este fenómeno es mucho menos famoso que la selección natural, pero es también un proceso importante detrás de la evolución de la biodiversidad. Para empezar, no es exclusivo del ADN no codificante. Ciertas características de los organismos que pensaríamos existen porque fueron favorecidas por la selección natural, en realidad pueden explicarse por deriva génica. Pero esa es otra de las tantas tangentes que no contaré hoy. Mi punto ahora es decir que la deriva génica es relevante para entender cómo evolucionan las secuencias neutrales. Este detalle importa porque permitió formular modelos matemáticos que nos permiten obtener información del pasado a partir de la secuencia de ADN de individuos del presente.
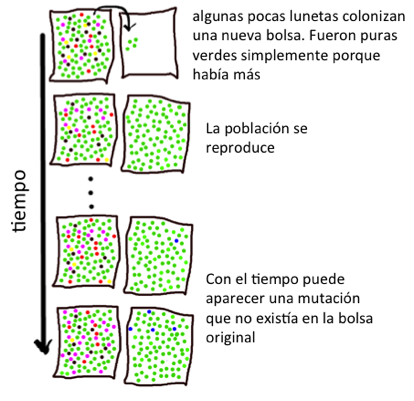
La lógica detrás de lo anterior no es tan complicada. Volvamos a las lunetas. Digamos que en una bolsa hay lunetas de varios colores, pero considerablemente muchas más verdes. Entonces es más probable que si tomo cuatro al azar todas sean verdes. Ahora imaginemos que las lunetas son individuos y que los colores representan diferentes secuencias de ADN neutral. Las lunetas verdes podrían ser un grupo que colonizó una isla y se aisló. Aunque su número se incremente a la misma cantidad que la bolsa original, el hecho de que en una bolsa existan varios colores y que en otra sólo verdes nos dice que la bolsa de sólo verdes puede provenir de una población que fue más pequeña recientemente y que la grande ha estado más estable. Esta es información del pasado que antes no sabíamos. Además, como ya dijimos, una secuencia de ADN puede mutar en una de sus letras. Por ende, gracias a que hay otras letras que no cambiaron, podemos deducir que la nueva secuencia (luneta azul, en la imagen) provino de la verde. Aunque la luneta azul no conozca a las lunetas rojas de la otra bolsa podemos decir que comparten un ancestro común. Esta es la lógica más básica detrás de la llamada teoría de coalescencia. El tic, toc del llamado reloj molecular entra en juego porque es posible conocer la tasa de mutación, es decir cada cuánto tiempo ocurre una. Así, la cantidad de diferencias entre dos secuencias se puede traducir en cuántos años han transcurrido desde que divergieron de un ancestro común.
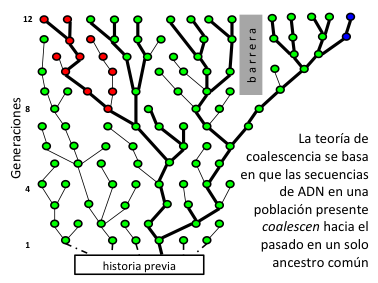
Simplifiqué mucho. El mundo biológico implica lunetas que migran, que se mezclan y cuyo ADN recombina, lo que complica los análisis de entretenidísima forma. La realidad metodológica detrás de estas ideas es un arduo tema de discusión. Mi intención al platicarles al respecto era sólo ponerlo como ejemplo introductorio para responder la pregunta del primer párrafo. La verdadera respuesta sería decir que el ADN puede utilizarse de muchas maneras para estudiar la biodiversidad y que en realidad sólo hemos empezado a explorar cómo. Ahí frente a nosotros se extiende el bosque de lo posible.
Acerca del autor
Alicia Mastretta Yanes es Bióloga egresada de la UNAM y actualmente cursa su doctorado en la University of East Anglia, Inglaterra. Su proyecto de doctorado explora la relación entre las características físicas del paisaje y la distribución de la diversidad genética en plantas de alta montaña de México.
Notas: * Está bien, cinco, si contamos al uracilo (U), pero ese existe en el ARN y no en el ADN, ya contaremos la historia.




